Biologen haben anhand von realen Messungen ein Computermodell entwickelt, mit dem sie genaue Prognosen machen können, wie oft Hefezellen in bestimmten Stresssituationen gewisse Gene ablesen. Dies ist ein wichtiger Schritt zu einem vertieften Verständnis von biologischen Systemen.
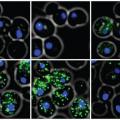
Setzt man Hefezellen einer Stresssituation aus, zum Beispiel indem man den Salzgehalt in ihrer Umgebung plötzlich erhöht, antworten sie darauf, indem sie bestimmte Gene aktivieren. Diese Gene sind an einen gewissen Signalweg gekoppelt, der im Fall des Salzstresses über ein Protein, den Transkriptionsfaktor Hog1, gestartet wird. Steigt die Salzkonzentration ausserhalb der Zellen an, wird Hog1 in die Zelle an den Ort transportiert, wo die Gene abgelesen werden, nämlich den Zellkern. Dort schaltet dieser Transkriptionsfaktor bestimmte Gene an, welche Proteine codieren und die Hefezellen befähigen, mit dem Salzstress umzugehen.
Nun könnte man annehmen, dass die genetische Antwort – sprich die Transkription der Salzstress-Gene – in einer Population von Hefezellen einheitlich ausfällt. Dem ist allerdings nicht so. Denn in einer Population «rauscht» es beträchtlich, was nichts anderes heisst, als dass sich jede einzelne Zelle zu einem bestimmten Zeitpunkt in einem unterschiedlichen Zustand befindet. So kann es etwa sein, dass in einer Zelle die Erbsubstanz DNS noch in einer kompakten, kondensierten Form vorliegt, sodass die für die Stressantwort gebrauchten Gene nicht sofort abgelesen werden können. Eine andere Zelle wiederum besitzt vielleicht eine zu geringe Konzentration von Hog1, um den Signalweg überhaupt in Gang setzen zu können.
Dieses zufällige «Rauschen» macht es sehr schwer vorauszusagen, wie die Population quantitativ auf den Stress reagieren wird. Für die Systembiologie ist es allerdings zentral, die komplexe und stochastische Dynamik der Genregulation zu verstehen und (quantitative) Voraussagen treffen zu können. Von solchen Prognosen erhoffen sich Systembiologen grundlegende Erkenntnisse über biologische Vorgänge.
Neues Modell anhand der Genantwort erstellt
Diese Wissenslücke füllt nun ein internationales Team von Systembiologen unter der Federführung von Mustafa Khammash, Professor für Regelungstheorie und Systembiologie am Departement Biosysteme der ETH Zürich in Basel und von Alexander van Oudenaarden vom MIT in Boston. Sie haben ein neues mathematisches Modell entwickelt, das diese Dynamik quantitativ abbildet. Es kann genau vorhersagen, wie die Hefezellen auf den Salzstress reagieren. Das Modell kann aber auch für andere Signalwege verwendet werden. Die Arbeit der beiden Forschungsgruppen wurde soeben in der Fachzeitschrift «Science» veröffentlicht.
Grundlage des Modells sind Laborexperimente, bei denen Hefezellen einer erhöhten Salzkonzentration im Nährmedium ausgesetzt wurden und dann die Konzentration der Boten-RNS des Gens STL1 gemessen wurden. Das von diesem Gen codierte Protein ist Teil der Signalkette bei Salzstress. Dessen Transkription wird durch das Protein Hog1 ausgelöst, aber nur dann, wenn Hog1 einen bestimmten Schwellenwert erreicht. Die Boten-RNS ist im Prinzip das spiegelverkehrte Abbild des genetischen Codes, wie er in der DNS festgelegt ist. Sie wird aus dem Zellkern ausgeschleust und dient im Zellplasma als Vorlage für den Zusammenbau des entsprechenden Proteins.
Computer wählt bestes Modell aus
Anhand dieser Daten untersuchte das Team von Mustafa Khammash eine Vielzahl mathematischer Modelle, um die Konzentration der STL1-Boten-RNS bei Salzstress vorauszusagen. Sie prüften alle Computermodelle, um schliesslich diejenigen auszusortieren, die entweder zu einfach waren, um zutreffende Voraussagen zu treffen, oder so kompliziert, dass sie Informationen abbildeten, die nicht in den Daten waren.
Das Modell, das die Konzentration von STL1-Boten-RNS am besten voraussagen konnte, wurde auf Herz und Nieren geprüft. Die Forscher errechneten damit die Konzentrationen von drei weiteren Boten-RNS-Molekülen, welche Hefezellen ebenfalls als Antwort auf den Salzstress bilden. Die errechneten Werte wurden schliesslich mit experimentellen Messungen verglichen, um so die Aussagekraft des Modells zu prüfen.
Mächtiger Ansatz
In dieser Arbeit vereinten die Forscher die Technologien aus zwei unterschiedlichen Forschungszweigen: die Messung von Konzentrationen einzelner Boten-RNS-Moleküle mit hoher zeitlicher Auflösung und eine von Khammashs Gruppe entwickelte Methode zur Analyse von Zufallsprozessen, die quantitativ beschreiben kann, wie sich Boten-RNS-Konzentrationen in Zellen dynamisch entwickeln und verteilen.
«Wir haben die beiden Werkzeuge dazu verwendet, um die Dynamik der Genregulierung in Zellen zu verstehen, die sich durch stochastische Unsicherheiten auszeichnen», sagt Khammash. Durch das Abhören des Zellrauschens mit diesen Methoden konnten die Forscher detaillierte Informationen der zugrunde liegenden Gen-Transkription gewinnen. Dadurch erreichten sie ein besseres Verständnis dieses fundamental wichtigen biologischen Prozesses. «Das zeigt die Mächtigkeit des systembiologischen Forschungsansatzes für die Biologie», sagt der ETH-Professor.
Literaturhinweis
Neuert G, Munsky B, Tan RZ, Teytelman L, Khammash M, van Oudenaarden A. Systematic Identification of Signal-Activated Stochastic Gene Regulation. Science 2013. 6119: 584, doi: 10.1126/science.1231456